Consensus CDSの領域をGRangesオブジェクトにする
Bioconductor - TxDb.Hsapiens.UCSC.hg19.knownGeneは便利なのですが、ある遺伝子に対応するtranscriptが多すぎてどうしたものかと困ってました。
で、UCSCのゲノムブラウザで見るとConsensus CDS(CCDS)のアノテーションもあるのですが、このBioconductorのパッケージからそれを取得する方法が分かりませんでした。
正確にはmakeTranscriptDbFromUCSC(genome="hg19", tablename="ccdsGene")などとしてCCDSの領域は取得はできるんですが、他のアノテーション(Gene IDなど)が一切消えるので全く使いものにならない感じです。
仕方がないのでCCDS ProjectのFTPサーバから元データを引っ張ってきてそれをパースしてアノテーションのあるCCDSのデータを作ったのでちょろっと共有しておきます。
まずはCCDS ProjectのFTPサーバからCCDSの領域とアノテーションを収めたファイルを取得します (ftp://ftp.ncbi.nlm.nih.gov/pub/CCDS/)。 reference genomeのビルドによっていくつかありますので、自分の目的に合ったものを取得しましょう。 私はftp://ftp.ncbi.nlm.nih.gov/pub/CCDS/archive/Hs37.3/CCDS.current.txtを取りました。
次にこのデータをRがパースしやすいようにPerlで整形します。元データはこんな感じのでデータになってます。
CCDS.current.txt:
#chromosome nc_accession gene gene_id ccds_id ccds_status cds_strand cds_from cds_to cds_locations match_type 1 NC_000001.8 LINC00115 79854 CCDS1.1 Withdrawn - 801942 802433 [801942-802433] Identical 1 NC_000001.10 SAMD11 148398 CCDS2.2 Public + 861321 879532 [861321-861392, 865534-865715, 866418-866468, 871151-871275, 874419-874508, 874654-874839, 876523-876685, 877515-877630, 877789-877867, 877938-878437, 878632-878756, 879077-879187, 879287-879532] Identical 1 NC_000001.10 NOC2L 26155 CCDS3.1 Public - 880073 894619 [880073-880179, 880436-880525, 880897-881032, 881552-881665, 881781-881924, 883510-883611, 883869-883982, 886506-886617, 887379-887518, 887791-887979, 888554-888667, 889161-889271, 889383-889461, 891302-891392, 891474-891594, 892273-892404, 892478-892652, 894308-894460, 894594-894619] Identical 1 NC_000001.10 PLEKHN1 84069 CCDS4.1 Public + 901911 909954 [901911-901993, 902083-902182, 905656-905802, 905900-905980, 906065-906137, 906258-906385, 906492-906587, 906703-906783, 907454-907529, 907667-907803, 908240-908389, 908565-908705, 908879-909019, 909212-909430, 909695-909743, 909821-909954] Identical 1 NC_000001.10 HES4 57801 CCDS5.1 Public - 934438 935352 [934438-934811, 934905-934992, 935071-935166, 935245-935352] Identical 1 NC_000001.10 ISG15 9636 CCDS6.1 Public + 948953 949857 [948953-948955, 949363-949857] Identical 1 NC_000001.10 C1orf159 54991 CCDS7.2 Public - 1018272 1026922 [1018272-1018366, 1019732-1019762, 1019860-1019885, 1021257-1021391, 1022518-1022583, 1022881-1022976, 1025732-1025807, 1026851-1026922] Identical 1 NC_000001.10 TTLL10 254173 CCDS8.1 Public + 1115433 1120521 [1115433-1115719, 1115862-1115980, 1116110-1116239, 1117120-1117194, 1117740-1117825, 1118255-1118426, 1119299-1119470, 1120348-1120521] Identical 1 NC_000001.10 TNFRSF18 8784 CCDS9.1 Public - 1138970 1141950 [1138970-1139339, 1139778-1139865, 1140749-1140871, 1141764-1141950] Identical
CDS領域の範囲が一行に収められてます。 RマスターならRでもサクッといけるんでしょうが、私は以下のPerlスクリプトで事前にTSVファイルに変換しました。 (追記: CCDSの入力ファイルが0-based coordinateだったので1-basedに修正)
parse_ccds.pl:
#!/usr/bin/env perl use v5.10; use strict; use warnings; say "chromosome\tnc_accession\tgene\tgene_id\tccds_id\tccds_status\tcds_strand\tcds_from\tcds_to\tmatch_type\tstart\tstop"; while (<>) { next if /^#/; chomp; my ($chromosome, $nc_accession, $gene, $gene_id, $ccds_id, $ccds_status, $cds_strand, $cds_from, $cds_to, $cds_locations, $match_type) = split /\t/; my @cds_ranges = map { my ($start, $stop) = split /-/; {start => $start+1, stop => $stop+1} } (split /,\s/, (substr $cds_locations, 1, -1)); foreach (@cds_ranges) { say "$chromosome\t$nc_accession\t$gene\t$gene_id\t$ccds_id\t$ccds_status\t$cds_strand\t$cds_from\t$cds_to\t$match_type\t$_->{start}\t$_->{stop}"; } }
そして、tsvファイルを吐きます。
cat CCDS.current.txt | perl parse_ccds.pl > CCDS.current.tsv
あとはRで読み込むだけです。幸い、makeGRangesFromDataFrameを使うことで、読み込んだデータフレームを簡単にGRangesオブジェクトに変換できます。
> library(GenomicRanges)
> ccds = read.csv("CCDS.current.tsv", sep="\t")
> ccds = makeGRangesFromDataFrame(ccds, strand.field="cds_strand", keep.extra.columns=T)
> ccds
GRanges object with 287116 ranges and 8 metadata columns:
seqnames ranges strand | nc_accession gene gene_id ccds_id ccds_status cds_from cds_to
<Rle> <IRanges> <Rle> | <factor> <factor> <integer> <factor> <factor> <integer> <integer>
[1] 1 [801942, 802433] - | NC_000001.8 LINC00115 79854 CCDS1.1 Withdrawn 801942 802433
[2] 1 [861321, 861392] + | NC_000001.10 SAMD11 148398 CCDS2.2 Public 861321 879532
[3] 1 [865534, 865715] + | NC_000001.10 SAMD11 148398 CCDS2.2 Public 861321 879532
[4] 1 [866418, 866468] + | NC_000001.10 SAMD11 148398 CCDS2.2 Public 861321 879532
[5] 1 [871151, 871275] + | NC_000001.10 SAMD11 148398 CCDS2.2 Public 861321 879532
... ... ... ... ... ... ... ... ... ... ... ...
[287112] Y [16168463, 16168738] + | NC_000024.9 VCY1B 353513 CCDS56618.1 Public 16168169 16168738
[287113] Y [16835028, 16835148] + | NC_000024.9 NLGN4Y 22829 CCDS56619.1 Public 16835028 16953141
[287114] Y [16936067, 16936252] + | NC_000024.9 NLGN4Y 22829 CCDS56619.1 Public 16835028 16953141
[287115] Y [16941609, 16942398] + | NC_000024.9 NLGN4Y 22829 CCDS56619.1 Public 16835028 16953141
[287116] Y [16952292, 16953141] + | NC_000024.9 NLGN4Y 22829 CCDS56619.1 Public 16835028 16953141
match_type
<factor>
[1] Identical
[2] Identical
[3] Identical
[4] Identical
[5] Identical
... ...
[287112] Identical
[287113] Identical
[287114] Identical
[287115] Identical
[287116] Identical
-------
seqinfo: 24 sequences from an unspecified genome; no seqlengths
遺伝子毎のCDS領域を得たければ、Gene IDのアノテーションがccdsにありますのでsplitでGRangesListオブジェクトにできます。
> ccdsbygene = split(ccds.public, ccds.public$gene_id)
> ccdsbygene
GRangesList object of length 18383:
$1
GRanges object with 8 ranges and 8 metadata columns:
seqnames ranges strand | nc_accession gene gene_id ccds_id ccds_status cds_from cds_to
<Rle> <IRanges> <Rle> | <factor> <factor> <integer> <factor> <factor> <integer> <integer>
[1] 19 [58858387, 58858394] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[2] 19 [58858718, 58859005] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[3] 19 [58861735, 58862016] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[4] 19 [58862756, 58863052] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[5] 19 [58863648, 58863920] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[6] 19 [58864293, 58864562] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[7] 19 [58864657, 58864692] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
[8] 19 [58864769, 58864802] - | NC_000019.9 A1BG 1 CCDS12976.1 Public 58858387 58864802
match_type
<factor>
[1] Identical
[2] Identical
[3] Identical
[4] Identical
[5] Identical
[6] Identical
[7] Identical
[8] Identical
...
<18382 more elements>
-------
seqinfo: 24 sequences from an unspecified genome; no seqlengths
RStudioから使えるR Markdownで日本語のPDFを作成する
RStudioではRの実行結果を埋め込んでドキュメントやレポートを作成するのにLaTeXベースのものとMarkdown(の亜種のR Markdown)ベースのものが選べるのですが、R Markdownから日本語を含んだレポートをPDF化するのに手間取ったのでその方法を記述します。 私の環境はMac OS X Mavericksですが、他の環境でも共通する点はあると思います。
準備
まずMarkdownをLaTeXにするPandocとLaTeXの処理系(XeLaTeX)が必要です。
恐らくPandocに関してはRStudioに同封されているので問題無いと思います。
LaTeXの処理系に関してはMacTeXを利用するのが最も手早いです。しかし、2GB以上もある巨大なファイルをダウンロードし、インストールする羽目になるので、コアだけを集めたBasicTeXでも十分です。
BasicTeXをインストールしたら、いくつか必要なパッケージをインストールします1:
sudo tlmgr update --self --all sudo tlmgr install framed sudo tlmgr install titling sudo tlmgr install collection-langjapanese
設定
続いてRStudioを起動し、必要な設定を行います。
“RStudio” ➠ “Preferences…” ➠ “Sweave”と選択し、取り敢えずデフォルトをの設定を以下のようにします。
- Weave Rnw files using: knitr
- Typeset LaTeX into PDF using: XeLaTeX
- Preview PDF after compile using: System Viewer
最後の設定はRStudioのPDFビューアーが気に入らなかっただけで、私の好みです(PDF Reader Xを使っています)。
また、この設定はデフォルトのもので、プロジェクトでは別個に設定する必要があります。

R Markdownファイルの作成
“File” ➠ “New File” ➠ “R Markdown…” と選択し、適当にタイトルを設定してR Mawkdownのひな形作成します。ここでは、タイトルは「日本語PDF作成」としました。

しかし、これだけではプロジェクトの設定が反映され、先ほどのデフォルト設定が無視されてしまう場合があります。 そこで、ファイルの先頭を以下のように変更します。
変更前:
--- title: “日本語PDF作成” output: pdf_document ---
変更後:
---
title: “日本語PDF作成"
output:
pdf_document:
latex_engine: xelatex
mainfont: Hiragino Kaku Gothic Pro
---
latex_engineとしてxelatexを設定することで、UTF8などのエンコーディングに対応できるようになり、mainfontを設定することで、出来上がったPDFファイルの日本語フォントが表示されなくなる問題を修正しています。
ここでは、Hiragino Kaku Gothic Proを使いましたが、MacのFont Bookから、日本語に対応した好きなフォントを選ぶこともできます。
最後に日本語を含むR Markdownを書き、”Knit PDF”をクリックするとPDFが表示されます。

参考
JuliaTokyo #2 でモンテカルロ法について話してきました
9月27日の JuliaTokyo #2 で『Juliaで学ぶ Hamiltonian Monte Carlo (NUTS 入り)』と題してHamiltonian Monte Carlo (HMC) とStanで使われているNUTSについてお話してきました。
内容
サンプルコード: bicycle1885/JuliaTokyo2HMC · GitHub
MCMC自体については30分ではほとんど説明できませんでしたが、会場のほとんどの方が既に知っているようでしたのでちょうど良かったようです。
全体の流れとしては、Metropolis-Hastings、Hamiltonian Monte Carlo、No-U-Turn Samplerの順に3つサンプラーを実装コードと正規分布からのサンプリングを例に紹介し、どのように前のサンプラーの弱点を克服していってるかを説明しました。
Unicodeプログラミング
サンプルコードはJuliaの愉しみのひとつであるUnicode文字をふんだんに使っていたところが面白いところです。
function build_tree(L::Function, ∇L::Function, θ::Vector{Float64}, r::Vector{Float64}, u::Float64, v::Int, j::Int, ϵ::Float64)
if j == 0
θ′, r′ = leapfrog(∇L, θ, r, v * ϵ)
C′ = u ≤ exp(L(θ′) - r′ ⋅ r′ / 2) ? Set([(θ′, r′)]) : Set([])
s′ = int(L(θ′) - r′ ⋅ r′ / 2 > log(u) - Δmax)
return θ′, r′, θ′, r′, C′, s′
else
θ⁻, r⁻, θ⁺, r⁺, C′, s′ = build_tree(L, ∇L, θ, r, u, v, j - 1, ϵ)
if v == -1
θ⁻, r⁻, _, _, C″, s″ = build_tree(L, ∇L, θ⁻, r⁻, u, v, j - 1, ϵ)
else
_, _, θ⁺, r⁺, C″, s″ = build_tree(L, ∇L, θ⁺, r⁺, u, v, j - 1, ϵ)
end
s′ = s′ * s″ * ((θ⁺ - θ⁻) ⋅ r⁻ ≥ 0) * ((θ⁺ - θ⁻) ⋅ r⁺ ≥ 0)
C′ = C′ ∪ C″
return θ⁻, r⁻, θ⁺, r⁺, C′, s′
end
end
ちなみにjulia-vimはlatex記法サポートしてます https://t.co/iT48OQLi6r #JuliaTokyo
— 佐藤ヾ(⌒(「 'ω')「ガオー建太 (@bicycle1885) 2014, 9月 27箴言
Knuth 「尚早な最適化は諸悪の根源」
M. Jackson 「最適化第一法則: 最適化するな, 第二法則: まだするな」
#JuliaTokyo
— 佐藤ヾ(⌒(「 'ω')「ガオー建太 (@bicycle1885) 2014, 9月 27+α
俺のRandomForests.jlが某巨大企業で使われているらしい!凄い! #JuliaTokyo
— 佐藤ヾ(⌒(「 'ω')「ガオー建太 (@bicycle1885) 2014, 9月 27社内で改造しているようですが、手続き上のアレでプルリクは受けられないようです(´・ω・`)
コマンドを並列に実行するGNU parallelがとても便利
最近のコンピュータは複数のCPUコアを持っているので並列にコマンドを実行することができます。 たくさんの同じようなファイルに同じ処理を実行することは、私のやっているバイオインフォマティクスではよくあります。
しかし自分で並列に実行するスクリプトを書くことはそれほど簡単ではなく、ログや実行結果の確認など煩雑な処理を書かなければいけません。 この記事では、そうした処理を簡単にするGNU parallelというツールを紹介します。

GNU parallel
UNIX系のOSではインストールはとても簡単です。MacでしたらHomebrewを使って、Linuxでは各ディストリビューションのパッケージマネージャからインストールできます。 詳しくはGNU parallelのウェブページを参照して下さい(http://www.gnu.org/software/parallel/)。
Homebrew:
brew install parallel
パッケージマネージャが使えない環境でも、ソースコードからビルドすることができます。 ダウンロードページ(http://ftp.gnu.org/gnu/parallel/)から、最新のparallel(parallel-latest.tar.bz2)をダウンロードして展開し、ビルドしましょう。 必要ならばconfigureに--prefixも渡せます。
Source:
./configure && make && make install
なお、最初にparallelコマンドを実行する際に引用のお願いメッセージが出ますが、これはparallel --bibtexを一度実行すれば抑えられます。
Hello, GNU parallel !
インストールが終わったら、早速GNU parallelを実行してみましょう。
~/t/sample $ parallel echo ::: hello world ! hello ! world
GNU parallelのparallelコマンドは、実行するコマンドと、そのコマンドに適用するいくつかの引数を引数としてとります。
上の例では、echoが実行するコマンドで、:::の後に続く3つの引数hello, world, !がそれぞれechoコマンドに適用する引数です。
echoされたメッセージが元の引数の順番と一致していないことから分かる通り、echoコマンドは並列に実行されるため、各引数の実行順は不定になります。
:::は複数つなげることで、引数の組み合わせの積をつくることもできます。
~/t/sample $ parallel echo ::: hello, bye, ::: Alice Bob Charlie ::: ! hello, Alice ! hello, Bob ! hello, Charlie ! bye, Alice ! bye, Bob ! bye, Charlie !
ファイルや標準入出力から引数を与える
以下のようなファイル名のリストを収めたファイル(list.txt)があるとします。
~/t/sample $ cat list.txt foo.txt bar.txt baz.txt
-a <file>オプションで、ファイルの各行を引数としてコマンドを並列に走らせることができます。
list.txtの各ファイルを並列に圧縮するには以下のようにします。
~/t/sample $ ls bar.txt baz.txt foo.txt list.txt ~/t/sample $ parallel -a list.txt gzip ~/t/sample $ ls bar.txt.gz baz.txt.gz foo.txt.gz list.txt
パイプで引数を渡すこともできます。その時は-a -とオプションを渡します。
~/t/sample $ cat list.txt | parallel -a - gzip ~/t/sample $ ls bar.txt.gz baz.txt.gz foo.txt.gz list.txt
引数の区切り文字はデフォルトでは\n(newline)です。
-0(--null)オプションで区切り文字が\0(null)文字になります。
他にも、<や::::を使って引数の納められたファイルを渡すことができます。
~/t/sample $ parallel gzip < list.txt ~/t/sample $ parallel gzip :::: list.txt
引数の置き換え
コマンドに渡す引数の位置が最後でない場合や、特別な処理をしたい場合に引数の置換え位置を指定するをする必要があります。
その際には{}を使います。
~/t/sample $ parallel 'find {} -name "README*"' ::: ~/vendor/julia ~/vendor/vector
/Users/kenta/vendor/vector//old-testsuite/microsuite/README
/Users/kenta/vendor/vector//README.md
/Users/kenta/vendor/julia//contrib/mac/app/README
/Users/kenta/vendor/julia//contrib/README.ackrc.txt
...
さらに、{.}でファイル名の拡張子を除いた引数にしたり、{/}でファイル名だけ取り出したりできます。
~/t/sample $ parallel 'echo {.}' ::: tmp/foo.txt.gz tmp/bar.txt.gz
tmp/foo.txt
tmp/bar.txt
~/t/sample $ parallel 'echo {/}' ::: tmp/foo.txt.gz tmp/bar.txt.gz
foo.txt.gz
bar.txt.gz
実行されるコマンドの確認
--dry-runをつかえば、引数がどのようにコマンドに適用されてどのようなコマンドが実際に走るかを確認できます。
実際に実行する前に一度確認すると良いでしょう。
~/t/sample $ cat list.txt | parallel -a - --dry-run gzip gzip foo.txt gzip bar.txt gzip baz.txt
実行結果の取得
標準出力と標準エラーに吐かれた結果をファイルとして取得することも容易です。
--results <outputdir>とすることで出力を各引数毎にディレクトリに構造化して保存できます。
~/t/sample $ parallel --results results 'perl -E "say STDOUT \"stdout\"; say STDERR \"stderr\""' ::: A B C
stdout
stderr
stdout
stderr
stdout
stderr
~/t/sample $ tree results/
results/
└── 1
├── A
│ ├── stderr
│ └── stdout
├── B
│ ├── stderr
│ └── stdout
└── C
├── stderr
└── stdout
4 directories, 6 files
~/t/sample $ cat results/1/A/stderr
stderr
~/t/sample $ cat results/1/A/stdout
stdout
実行結果の確認
時間のかかるコマンドや引数が多い場合など、実行結果の確認が大変な場合があります。
そこで、--joblog <logfile>を使えばコマンドのexit statusが確認しやすくなります。
~/t/sample $ parallel --joblog joblog.txt 'sleep {}; exit {}' ::: 0 1 2 3
~/t/sample $ cat joblog.txt
Seq Host Starttime JobRuntime Send Receive Exitval Signal Command
1 : 1407646480.280 0.072 0 0 0 0 sleep 0; exit 0
2 : 1407646480.284 1.144 0 0 1 0 sleep 1; exit 1
3 : 1407646480.289 2.125 0 0 2 0 sleep 2; exit 2
4 : 1407646480.294 3.153 0 0 3 0 sleep 3; exit 3
一目見て分かる通り、実行時間や実際に実行されたコマンドなども記録されるため、問題が発生した時のトラブルシューティングに非常に役に立ちます。 また、終わったコマンドから順に追記されていくため、進捗状況の確認もできます。
並列ジョブの制御
ファイルのダウンロードなどを並列に行いたいが過剰に負荷を掛けたくない場合や、逆にCPU boundでない処理をCPUのコア数より多く並列させたいときなどに同時実行するジョブ数を指定できると便利です。
デフォルトでは、マシンのコア数分だけ並列させるため、共有サーバーなどではCPUを占拠してしまい迷惑になることも考えられます。
そのようなときは--jobs <N>(-j <N>)オプションを使って、
以下のように6つの引数を1, 2, 3, 6並列でそれぞれ実行して実行時間を見てみましょう。
~/t/sample $ time parallel --jobs 1 'sleep {}' ::: 1 1 1 1 1 1
7.06 real 0.26 user 0.10 sys
~/t/sample $ time parallel --jobs 2 'sleep {}' ::: 1 1 1 1 1 1
3.58 real 0.27 user 0.11 sys
~/t/sample $ time parallel --jobs 3 'sleep {}' ::: 1 1 1 1 1 1
2.44 real 0.28 user 0.11 sys
~/t/sample $ time parallel --jobs 6 'sleep {}' ::: 1 1 1 1 1 1
1.30 real 0.30 user 0.12 sys
--jobs 50%などと書くことで全体の半分のCPUコアを使う指定もできます。
~/t/sample $ time parallel --jobs 50% 'sleep {}' ::: 1 1 1 1 1 1
3.59 real 0.26 user 0.11 sys
また、メモリを沢山必要とする計算などでは、--noswapオプションを指定することで、メモリのスワップが発生している時には新しいジョブを実行しないようにすることもできます。
他の便利そうな機能
ここで紹介した機能はGNU parallelのごく一部で、他にも有用なオプションなどがたくさんあります。
一度、man parallel_tutorialやman parallelに目を通すことをオススメします。
私はまだ実際に実行して確認していませんが、GNU parallelにはSSH越しに複数のノードで並列にジョブを実行させる機能やパイプライン処理で中間のボトルネックになっている処理だけ並列化するなどの機能もあるようです。
これらの機能は確認し次第、ブログに追記していこうと思います。
statisticsにt検定を実装しました
ちょっとHaskellのstatisticsパッケージを見ていたら、統計的仮説検定に何故かt検定が無かったのでチョイチョイと実装しました。
https://github.com/bos/statistics/pull/66
masterの一歩手前のconfidenceブランチに取り込まれましたので、そのうち使えるようになると思います。
この実装で以下の3つの2標本検定が使えるようになります。
- Student's t-test
- Welch's t-test
- paired samples t-test
詳細については、Wikipediaを参照下さい。
statisticsパッケージの検定のAPIがに統一感がなくてイケてない感じがするので、なにか思いついたらそちらも修正したいです。あとp値や統計量が得られないのはさすがに良くない気がします。
JuliaTokyo #1 を開催しました
日本で(多分)最初のJuliaの勉強会 JuliaTokyo #1 を開催しました。イベントページはこちらです。 http://juliatokyo.connpass.com/event/6891/
当日は40人ほどの参加者が集まり、いつの間にJuliaがそんなに一般化したんだという感じでしたが、Julia歴1週間とか当日の朝から始めましたというように、何処からかJuliaが熱いらしいとの噂を聞きつけてJuliaを使ってみようという新しいもの好きの人たちが多い印象でした。
私はパッケージの作り方についてお話をしてきた次第です。
以下は、それぞれの発表と私のひとこと感想です。
メインセッション
Juliaのこれまでとこれから - @sorami
Juliaの背景的な情報やどんなエコシステムになっているかを紹介していました。 JuliaはPythonやRからユーザを取り込みつつ、共に進化していく道を辿るようです。
Julia100本ノック - @chezou
Juliaでnumpyの100 numpy exercisesを書いた話です。 Juliaのやり方とnumpyのやり方はベクトル化などの使い方がかなり違い、苦労があったようです。 あとJuliaのベクトル内包表記は速いのでガンガン使っていきましょう!
Juliaのパッケージを作ろう! - @bicycle1885
私の発表です。 Juliaのパッケージをどのようにつくるかを解説しました。 パッケージ名やディレクトリ構成などについて、ドキュメントに書いてない慣習などが大事だったりします。
ビジュアライズJulia - @nezuq
"The Grammar of Graphics"のlayerやscaleの概念や、Gadfly.jlとの関係の話でした。 可視化関係のライブラリはまだそれほどなので、今後充実してくると良いですね。
メカ女子将棋 - @kimrin
Juliaで将棋AIを作った話でした。やっぱりJuliaはCなどに比べ開発がずっとしやすいですね。 盤面の表現にはJuliaの128bit整数を使っているようです。
A Brief JuliaCon Report / Natural Language Processing with Julia - Pontus Stenetorpさん
https://github.com/JuliaCon/presentations/blob/master/JuliaNLP/JuliaNLP.pdf
先月Chicagoで開催されたJuliaConのレポートと、そのときに発表されたJuliaでの自然言語処理のお話をしていただきました。 JuliaConでは、アカデミックの人がメインだったものの、実際にサービスにJuliaを投入している会社もあったようです。 Juliaで自然言語のパーサallenを作って、パフォーマンスも満足いくものになったようです。
LT
Juliaでちゃんとしたコードを書く - 関さん
当日の朝からJuliaを始めて、どうするとJuliaが遅くなるかのお話でした。 マニュアルのperformance tipsマジ重要です。読みましょう!!
Julia0.3でランダムフォレスト - @gepuro
DecisionTree.jlを使ってみたようです。 DecisionTree.jlはID3を決定木構築のアルゴリズムに使っているので、CARTが使いたければ私のRandomForests.jlを使いましょう!
Plotly Julia API - Yasunobu Igarashiさん
Plotlyというデータの可視化や共有のためのプラットフォームの紹介でした。 JuliaのAPIもあるんですね、すごい!
LightTableを使いましょう - @QuantixResearch
LightTable + Jewel でJuliaを開発しようという話でした。 インラインで変数の値が見れたり、補完もかなり強力なようです。あとで使ってみよう!
感想
新しいおもちゃを手にしたときの子供のような、Juliaに対する期待と興奮が垣間見れたように思います。 おぉ!Juliaすごい!いいじゃん!というような熱気がありました。 Twitterでしか見たことない人と実際にお話したり、情報交換をしたりも大変有益でした。 反省点としては、ちょっと初心者向けの話が少なすぎたかなと思います。次回は初心者向けセッションを設けて欲しいとの要望が多かったです。 会場を用意していただいた株式会社ブレインパッドの皆様、本当にありがとうございました。
あと、本家Juliaステッカーをもらえたのがすごい嬉しかった!
本場シカゴからやって来たステッカー貰った(((o(*゚▽゚*)o))) pic.twitter.com/1AJeQCp0ml
— 佐藤ヾ(⌒(「 'ω')「ガオー建太 (@bicycle1885) 2014, 7月 5HaskellのFusionがあれば速度と抽象化を両立できる
データの分析をする際には配列やベクトルは欠かせないデータ構造です。 大体どの言語にも大体配列は用意されていて、そこにサンプルのデータ等を入れて統計量を計算したり関数に渡して回帰をしたりするわけです。 ベクトルという単位でデータの塊を扱うものの、実際のアルゴリズムではベクトル内の要素一つひとつを見ていって何か処理をしたり計算をすることが多いでしょう。 その際、命令型言語ではforループを陽に使って要素にアクセスすることになります。
簡単な例を見てみましょう。ベクトルv1とv2の内積を求める関数をC言語で書くと以下のようになります。
function dot(double* v1, double* v2, size_t n) { double s = 0.0; for (int i = 0; i < n; i++) { s += v1[i] * v2[i]; } return s; }
特に難しいところはありませんね:)
ベクトル化された記法
しかし、大きなベクトルに対してこうして陽にループを書くことは、PythonやRではある種の「悪手」であると認識されています。 理由は単純で、PythonやRのインタープリタのループが遅すぎるため、NumPyのようなCやFortranなどで書かれた高速なライブラリに配列(実際にはPythonのオブジェクトが持つメモリ上のバッファへのポインタ)を渡して計算させるのが定石になっています*1。
NumPyやSciPyではこうしたベクトルに対する関数や演算子が大量に用意されており、これらを組み合わせれば、明示的なforループを避けて簡潔に高速な計算ができるようになっているわけです。こうした操作をベクトル化(vectorized)された操作と言われています。
例えば、先程と同様2つのベクトルの内積を計算するならNumPyにnumpy.dot関数が予め用意されています。
import bumpy as np v1 = np.array([1.0, 2.0, 3.0]) v2 = np.array([2.0, 3.0, 4.0]) print(np.dot(v1, v2))
ベクトルの要素ごとの積(*)と総和を求める関数を使えば、内積計算を以下のようにも書くことができます。
def dot(v1, v2): return np.sum(v1 * v2)
分散の計算
こうしたベクトル化された関数を組み合わせて標本分散(不偏分散)を計算してみましょう。 数式での定義は以下のとおりです。
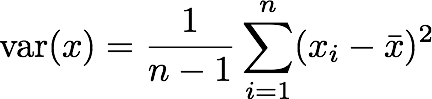
これを単純にNumPyを使ったPythonのコードに写してみましょう。(もちろん、NumPyには分散を計算するnp.var関数が用意されています!)
def var(x): return np.sum((x - np.mean(x))**2) / (np.size(x) - 1)
ベクトル化された関数を使えば、定義式をほとんどそのまま移すだけで、分散を計算する関数が作れました。
しかし、この方法には問題があります。それは、計算の途中に出てくる式の計算のために不要な一時配列が確保され、余計なメモリを食っていることです。
上の例では、x - np.mean(x)では、元の配列xと同じ大きさの配列が確保され、平均からの差分をそこに格納していく形になっています。(x - np.mean(x))**2ではさらに二乗の結果を格納する配列が確保されています。これらの一時的な配列は、本来分散を計算するためにはまったく不要なものです。
内積の例でも、np.sum(v1 * v2)のところでnp.sumに渡す一時ベクトルが新たに作られてしまっています。
def var(x): return np.sum((x - np.mean(x))**2) / (np.size(x) - 1) # ~~~~~~~~~~~~~~ # ^ # ~~~~~~~~~~~~~~~~~~~ # ^ # 余計なベクトルが2個作られている
Pythonのforループは遅くて使えないため、これを回避するためには、CやCythonで陽にループを書くハメになります。
#include <stdlib.h> #include <stdio.h> // Two-pass algorithm: // http://en.wikipedia.org/wiki/Algorithms_for_calculating_variance#Two-pass_algorithm double var(double* v, size_t n) { double mean = 0.0; for (int i = 0; i < n; i++) { mean += v[i]; } mean /= n; double ssd = 0.0; for (int i = 0; i < n; i++) { ssd += (v[i] - mean) * (v[i] - mean); } return ssd / (n - 1); }
先ほどのベクトル化された計算と比べると、ループに展開したコードはかなり読みにくく、定義式との対応がわかりづらくなってしまいました。
ちなみに、Juliaでは、マクロを使ってベクトル化された記法からループへと展開するライブラリDevectorize.jlがあります。
fusionを使って宣言的に書き、効率的なコードを吐く
宣言的なベクトル化された記法をしつつ、ループと同等の効率的な実行を可能にするのがfusionです。
fusionでは、中間にできてしまう一時的なベクトルをライブラリによる自動的なコードの書き換え規則とコンパイラによる最適化で排除し、効率的なコードを生成する技術です。
Haskellのvectorパッケージでは、ほとんど意識する必要なくベクトル計算のfusionを行ってくれます。
unboxedな値を格納するベクトルを使うなら、Data.Vector.Unboxedモジュールをインポートして使います。ここでは見た目を簡潔にするため、Preludeの名前が衝突する関数を隠しています。
import Prelude hiding (sum, zipWith) import Data.Vector.Unboxed dot :: Vector Double -> Vector Double -> Double dot v1 v2 = sum $ zipWith (*) v1 v2
v1 * v2ではなくzipWithを使ってる点が若干分かりにくいかもしれませんが、ループを書かずに宣言的に内積が定義できています。それでいて、実はこのdot関数は計算の中間データを保持するベクトルを作っていません!
fusionがどうやって動いているのか
実際にfusionがどのような仕組みになっているか簡単な例で見てみましょう。 ここでは、Stream Fusion - From Lists to Streams to Nothing at Allのサンプルの一部ををGHCで試せるように書いてみます。 これは、stream fusionというfusionの一例です。 vectorパッケージではVector型に対してfusionを行っていますが、ここではリストを使っています。
例えば、map f . map gというコードは関数合成の結合則とstream . unstream ≡ idという関係式から、
map f . map g ≡ (unstream . mapS f . stream) . (unstream . mapS g . stream)
≡ unstream . mapS f . (stream . unstream) . mapS g . stream
≡ ustream . mapS f . mapS g . stream
となります。ここで、streamとunstreamが打ち消しあうことで、リストとStreamの変換が消えますが、これはGHCのrewrite ruleによって実現しています。
さらに、GHCの最適化によりStepの生成も排除され、効率的なコードが吐かれるわけです。
リストに対するstream fusionのより詳しい実装は論文の著者等によるstream-fusionパッケージを参照してください。ただ、最近のGHCは既にリストののfusionをサポートしているため、上のコードやstream-fusionパッケージを使ったものは普通に書いたものと同等のパフォーマンスになりました*2。
分散の計算、再び
分散の計算をvectorパッケージを使って書いてみましょう。 fusionの効果を見るため、わざと関数を分けて定義しています。
import Prelude hiding (sum, length, zipWith, map) import System.Environment (getArgs) import Data.Vector.Unboxed as V -- | pairwise product (.*) :: Vector Double -> Vector Double -> Vector Double x .* y = zipWith (*) x y -- | sample mean mean :: Vector Double -> Double mean v = sum v / fromIntegral (length v) -- | deviations from the mean deviation :: Vector Double -> Vector Double deviation v = map (\x -> x - mean v) v -- | vectorized square square :: Vector Double -> Vector Double square v = v .* v -- | sum of squared deviations from the mean ssd :: Vector Double -> Double ssd = sum . square . deviation -- | unbiased sample variance var :: Vector Double -> Double var v = ssd v / fromIntegral (length v - 1)
このようにかなり宣言的にプログラムを書くことができ、それぞれの関数は単独で使うことも、他の場所で部品として使うこともできるなど再利用性も極めて高いです。Cの方だと可能なのはせいぜい平均値を計算するmean関数をくくりだすくらいでしょうか。
パフォーマンス比較
大きい配列に対してパフォーマンスを比較しみましょう。 長さ100,000,000の配列に対してCのループを使った実装とパフォーマンスを比べてみます。 使用したコードは以下のリンクにあります。 Gist - variance
Haskell: 0.49s user 0.35s system 99% cpu 0.839 total C: 0.48s user 0.33s system 98% cpu 0.815 total
Haskellの実装とCの実装とでほとんど同じ速度がでていますね。Haskellのコンパイルには-fllvmでLLVMのコードを吐くようにすると何割か速くなりました。
本当に一時データが割り当てられていないかも見てみましょう。 Doubleは8byteなので、長さ100,000,000の入力ベクトルに対して800,000,000バイト程度が必要になります。 以下のヒープに割り当てられた領域の大きさからも、不要な配列が割り当てられていないのが分かります。
% ./variance-hs $(cat n) +RTS -s -RTS
800,121,472 bytes allocated in the heap
10,104 bytes copied during GC
44,312 bytes maximum residency (1 sample(s))
21,224 bytes maximum slop
764 MB total memory in use (0 MB lost due to fragmentation)
Tot time (elapsed) Avg pause Max pause
Gen 0 1 colls, 0 par 0.00s 0.00s 0.0001s 0.0001s
Gen 1 1 colls, 0 par 0.00s 0.06s 0.0562s 0.0562s
INIT time 0.00s ( 0.00s elapsed)
MUT time 0.50s ( 0.72s elapsed)
GC time 0.00s ( 0.06s elapsed)
EXIT time 0.00s ( 0.06s elapsed)
Total time 0.50s ( 0.83s elapsed)
%GC time 0.0% (6.8% elapsed)
Alloc rate 1,610,816,342 bytes per MUT second
Productivity 100.0% of total user, 60.0% of total elapsed
新しいfusion
このようにfusionは強力なツールですが、今までのstream fusionにはいくつか限界があります。 最後に、こうした制約を取っ払うために発案された2つのfusionを紹介しましょう。 実装の詳細に関しては、私の能力の限界を超えるため、興味のある方は元の論文を参照してください。
Generalized Stream Fusion
2つのベクトルを結合して新しいベクトルを作りたいことはよくあります。 しかし、既存のstream fusionでは一度に1つづつしか値を取り出せないため、ベクトルを塊として扱うmemcpyのような効率的なコピーができません。 また、同じ理由で複数の値に一気に操作をするSIMD命令も使うことができません。
しかしGeneralized Stream Fusionでは素朴なStreamをより柔軟なBundleという単位にまとめることでこれらの問題を解決してくれます。 2つのベクトルの内積を計算するベンチマークでは、SSEを使うようにしたCのコードと同等かそれ以上の速度を出しています*3。
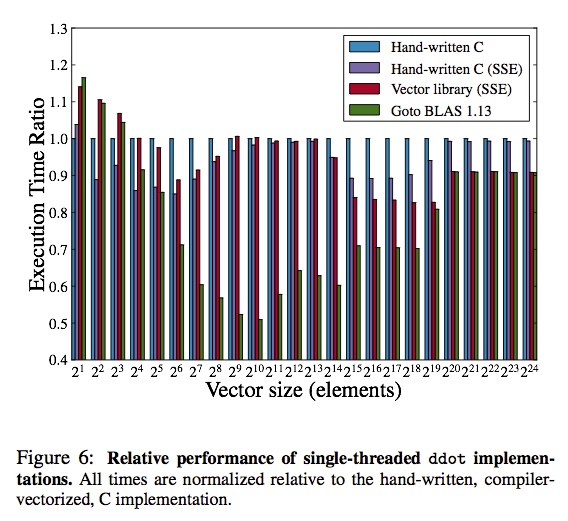
Generalized Stream Fusionでは、既存のHaskellのコードをほとんど変更することなく、合成可能な再利用性を保ったまま高速化できるのが魅力です。 この機能はvectorパッケージ上に実装されています。 現在OpenBLASやEigen3などの数値計算ライブラリと同等のパフォーマンスは出せていませんが、開発が進めばこれらのライブラリに迫るものができるかもしれません。
Data Flow Fusion
もう一つのstream fusionの拡張が、Data Flow Fusionです。 元のstream fusionではベクトルの値を変換して別のベクトルなり集計値を計算する「消費者」は常にひとりだけに限られています。 しかし、1つのベクトルから複数の統計量を計算したりする分岐をしたいことが常です。 大きなベクトルを複数回ループすることはキャッシュの有効利用ができないため、1つのループにまとめるのに比べて不利になってしまいます。
以下のコードは入力ベクトル(vec1)の要素をすべてインクリメントし(vec2)、正数のみを集め(vec3)たものとその中の最大の数をタプルにして返す関数です。
filterMax :: Vector Int -> (Vector Int, Int) filterMax vec1 = let vec2 = map (+ 1) vec1 vec3 = filter (> 0) vec2 n = foldl max 0 vec3 in (vec3, n)
この処理は一度のループで実行可能なはずですが、stream fusionでは1つのfusionにできない処理のようです。また、複数のベクトルに対して同時にループを行うときにループカウンタが重複してしまいレジスタを無駄遣いしてしまうこともパフォーマンスを悪化させてしまいます。
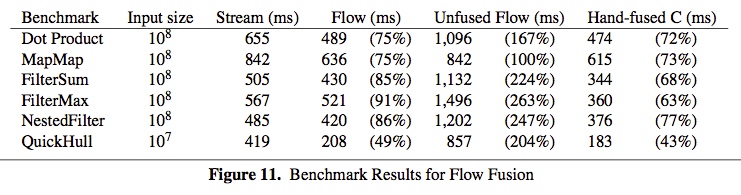
こうした問題をベクトル長の変化やデータフローを解析する高度なfusionによって解決するのがData Flow Fusionです*4。 実装は、Repaの配列に対するGHCの最適化プラグイン(repa-plugin)として提供されているようです。 この最適化により通常のstream fusion(Stream)に比べてdata flow fusion(Flow)では大きく性能が向上し、人間が手でfusionしたCのコード(Hand-fused C)に迫るパフォーマンスの向上を実現しています。
fusionは既に実用的な技術になっており、より強力になったfusionが使えるようになるのもそう遠くないかもしれません。